Manipulación de data frames usando tidyr
Última actualización: 2025-11-11 | Mejora esta página
Hoja de ruta
Preguntas
- ¿Cómo puedo cambiar el formato de los data frames?
Objetivos
- Entender los conceptos de formatos de datos largo y ancho y poder
convertirlos al otro formato usando
tidyr.
Las investigadoras a menudo quieren manipular sus datos del formato “ancho” al “largo”, o viceversa. El formato “largo” es donde:
- cada columna es una variable
- cada fila es una observación
En el formato “largo”, generalmente tienes una columna para la variable observada y las otras columnas son variables de ID.
Para el formato “ancho”, cada fila es un tema, por ejemplo un lugar o
un paciente. Tendrás múltiples variables de observación, que contienen
el mismo tipo de datos, para cada tema. Estas observaciones pueden ser
repetidas a lo largo del tiempo, o puede ser la observación de múltiples
variables (o una mezcla de ambos). Para algunas aplicaciones, es
preferible el formato “ancho”. Sin embargo, muchas de las funciones de
R han sido diseñadas para datos de formato “largo”. Este
tutorial te ayudará a transformar tus datos de manera eficiente,
independientemente del formato original.
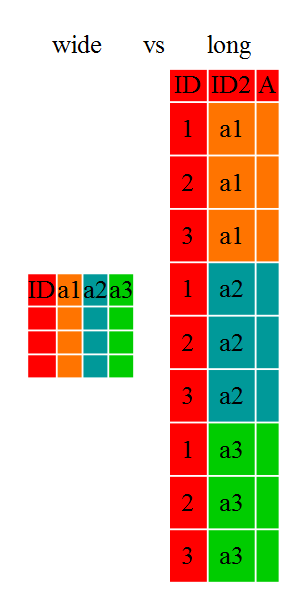
Estos formatos de datos afectan principalmente a la legibilidad. Para los humanos, el formato “ancho” es a menudo más intuitivo ya que podemos ver más de los datos en la pantalla debido a su forma. Sin embargo, el formato “largo” es más legible para las máquinas y está más cerca al formateo de las bases de datos. Las variables de ID en nuestros marcos de datos son similares a los campos en una base de datos y las variables observadas son como los valores de la base de datos.
Empecemos
Primero instala los paquetes necesarios, tidyr y
dplyr. Si aún no lo has hecho, puedes también instalar el
grupo de paquetes tidyverse que contiene varios paquetes
incluyendo tidyr y dplyr.
R
#install.packages("tidyr")
#install.packages("dplyr")
Ahora carga los paquetes usando library.
R
library("tidyr")
library("dplyr")
Primero, veamos la estructura structure del data frame gapminder:
R
str(gapminder)
SALIDA
'data.frame': 1704 obs. of 6 variables:
$ country : chr "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num 779 821 853 836 740 ...Desafío 1
¿Crees que el data frame gapminder tiene un formato largo, ancho o algo intermedio?
El data frame gapminder tiene un formato intermedio. No es
completamente largo porque tiene múltiples observaciones por cada
variable
(pop,lifeExp,gdpPercap).
A veces tenemos múltiples tipos de observaciones, como con el
data frame gapminder. Entonces tendremos formatos de
datos mixtos entre “largo” y “ancho”. Nosotros tenemos 3 “variables de
identificación” (continente,país,
año) y 3 “variables de observación” (pop,
lifeExp,gdpPercap). Generalmente, es
preferible que los datos estén en este formato intermedio en la mayoría
de los casos, a pesar de no tener TODAS las observaciones en una sola
columna. Esto es por que las 3 variables de observación tienen unidades
diferentes (y cada una corresponde a una columna con su propio tipo de
datos).
A menudo queremos hacer operaciones matemáticas con valores que usen
las mismas unidades, esto facilita el uso de funciones en R, que a
menudo se basan en vectores. Por ejemplo, si utilizamos el formato largo
y calculamos un promedio de todos los los valores de población
pop, esperanza de vida lifeExp y el PIB
gdpPercap este resultado no tendría sentido, ya que
devolvería una incongruencia de valores con 3 unidades incompatibles. La
solución es que primero manipulamos los datos agrupando (ver la lección
sobre dplyr), o cambiamos la estructura del marco de datos.
Nota: Algunas funciones de gráficos en R (por ejemplo
con ggplot2) realmente funcionan mejor con los datos de
formato ancho.
Del formato ancho al largo con gather()
Hasta ahora, hemos estado utilizando el conjunto de datos gapminder original muy bien formateado, pero los datos “reales” (es decir, nuestros propios datos de investigación) nunca estarán tan bien organizados. Veamos un ejemplo con la versión de formato ancho del conjunto de datos gapminder.
Descarga la versión ancha de los datos de gapminder desde [aquí] (https://raw.githubusercontent.com/swcarpentry/r-novice-gapminder/gh-pages/_episodes_rmd/data/gapminder_wide.csv) y guarda el archivo csv en tu carpeta de datos.
Cargaremos el archivo de datos para verlo. Nota: no queremos que las
columnas de caracteres sean convertidas a factores, por lo que usamos el
argumento stringsAsFactors = FALSE para para deshabilitar
eso, más información en la ayuda ?read.csv ().
R
gap_wide <- read.csv("data/gapminder_wide.csv", stringsAsFactors = FALSE)
str(gap_wide)
SALIDA
'data.frame': 142 obs. of 38 variables:
$ continent : chr "Africa" "Africa" "Africa" "Africa" ...
$ country : chr "Algeria" "Angola" "Benin" "Botswana" ...
$ gdpPercap_1952: num 2449 3521 1063 851 543 ...
$ gdpPercap_1957: num 3014 3828 960 918 617 ...
$ gdpPercap_1962: num 2551 4269 949 984 723 ...
$ gdpPercap_1967: num 3247 5523 1036 1215 795 ...
$ gdpPercap_1972: num 4183 5473 1086 2264 855 ...
$ gdpPercap_1977: num 4910 3009 1029 3215 743 ...
$ gdpPercap_1982: num 5745 2757 1278 4551 807 ...
$ gdpPercap_1987: num 5681 2430 1226 6206 912 ...
$ gdpPercap_1992: num 5023 2628 1191 7954 932 ...
$ gdpPercap_1997: num 4797 2277 1233 8647 946 ...
$ gdpPercap_2002: num 5288 2773 1373 11004 1038 ...
$ gdpPercap_2007: num 6223 4797 1441 12570 1217 ...
$ lifeExp_1952 : num 43.1 30 38.2 47.6 32 ...
$ lifeExp_1957 : num 45.7 32 40.4 49.6 34.9 ...
$ lifeExp_1962 : num 48.3 34 42.6 51.5 37.8 ...
$ lifeExp_1967 : num 51.4 36 44.9 53.3 40.7 ...
$ lifeExp_1972 : num 54.5 37.9 47 56 43.6 ...
$ lifeExp_1977 : num 58 39.5 49.2 59.3 46.1 ...
$ lifeExp_1982 : num 61.4 39.9 50.9 61.5 48.1 ...
$ lifeExp_1987 : num 65.8 39.9 52.3 63.6 49.6 ...
$ lifeExp_1992 : num 67.7 40.6 53.9 62.7 50.3 ...
$ lifeExp_1997 : num 69.2 41 54.8 52.6 50.3 ...
$ lifeExp_2002 : num 71 41 54.4 46.6 50.6 ...
$ lifeExp_2007 : num 72.3 42.7 56.7 50.7 52.3 ...
$ pop_1952 : num 9279525 4232095 1738315 442308 4469979 ...
$ pop_1957 : num 10270856 4561361 1925173 474639 4713416 ...
$ pop_1962 : num 11000948 4826015 2151895 512764 4919632 ...
$ pop_1967 : num 12760499 5247469 2427334 553541 5127935 ...
$ pop_1972 : num 14760787 5894858 2761407 619351 5433886 ...
$ pop_1977 : num 17152804 6162675 3168267 781472 5889574 ...
$ pop_1982 : num 20033753 7016384 3641603 970347 6634596 ...
$ pop_1987 : num 23254956 7874230 4243788 1151184 7586551 ...
$ pop_1992 : num 26298373 8735988 4981671 1342614 8878303 ...
$ pop_1997 : num 29072015 9875024 6066080 1536536 10352843 ...
$ pop_2002 : int 31287142 10866106 7026113 1630347 12251209 7021078 15929988 4048013 8835739 614382 ...
$ pop_2007 : int 33333216 12420476 8078314 1639131 14326203 8390505 17696293 4369038 10238807 710960 ...
El primer paso es formatear los datos de ancho a largo. Usando el
paquete tidyr y la función gather() podemos
juntar las variables de observación en una sola variable.
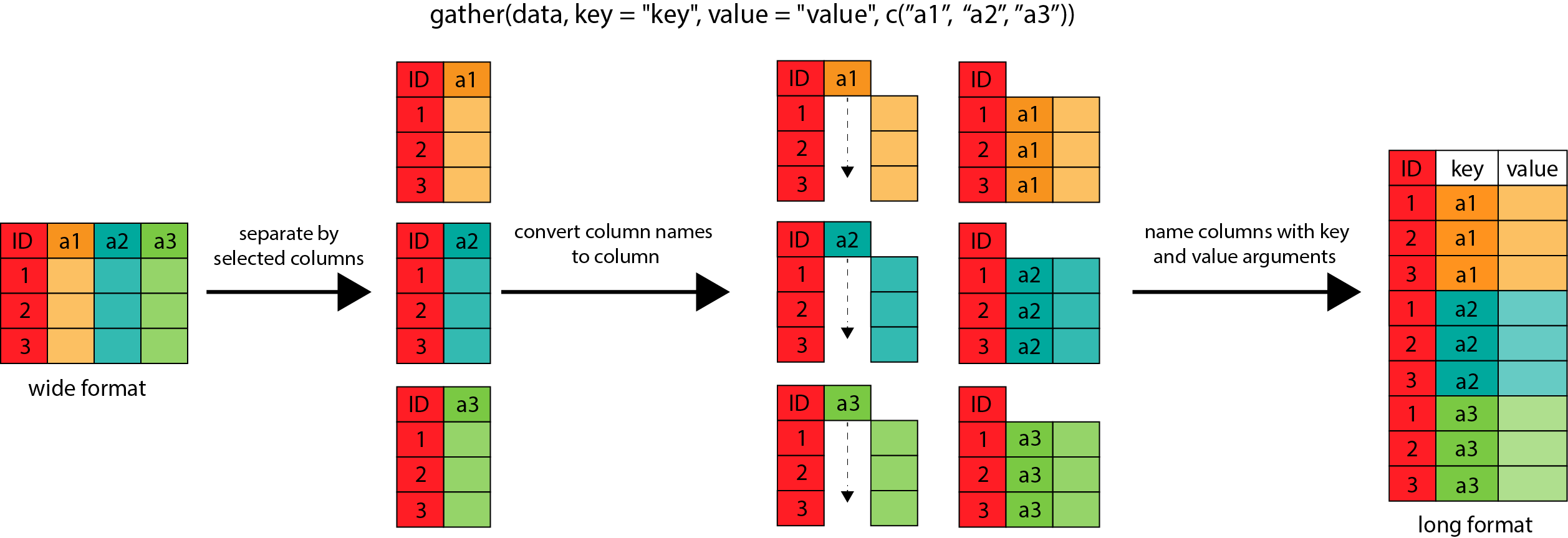
R
gap_long <- gap_wide %>%
gather(obstype_year, obs_values, starts_with("pop"),
starts_with("lifeExp"), starts_with("gdpPercap"))
str(gap_long)
SALIDA
'data.frame': 5112 obs. of 4 variables:
$ continent : chr "Africa" "Africa" "Africa" "Africa" ...
$ country : chr "Algeria" "Angola" "Benin" "Botswana" ...
$ obstype_year: chr "pop_1952" "pop_1952" "pop_1952" "pop_1952" ...
$ obs_values : num 9279525 4232095 1738315 442308 4469979 ...Aquí hemos utilizado la sintaxis con pipes (%>%)
igual a como lo que estábamos usando en el lección anterior con
dplyr. De hecho, estos son compatibles y puedes usar una
mezcla de las funciones tidyr y dplyr.
Dentro de gather(), primero nombramos la nueva columna
para la nueva variable de ID (obstype_year), el nombre de
la nueva variable de observación conjunta (obs_value),
luego los nombres de la antigua variable de observación. Nosotros
podríamos tener escritas todas las variables de observación, pero como
lo hacíamos en la función select() (ver la lección de
dplyr), podemos usar el argumento
starts_with() para seleccionar todas las variables que
comiencen con la cadena de caracteres deseada. Reunir o
gather también permite la sintaxis alternativa del uso
del símbolo - para identificar qué variables queremos
excluir (por ejemplo, las variables de identificación o
ID)
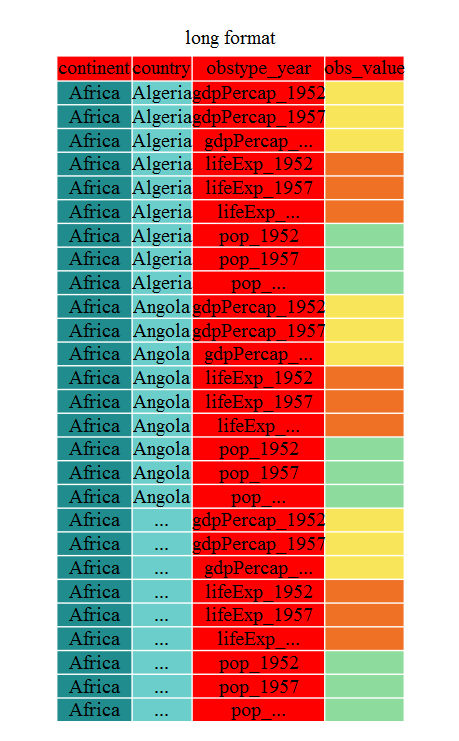
R
gap_long <- gap_wide %>% gather(obstype_year, obs_values, -continent, -country)
str(gap_long)
SALIDA
'data.frame': 5112 obs. of 4 variables:
$ continent : chr "Africa" "Africa" "Africa" "Africa" ...
$ country : chr "Algeria" "Angola" "Benin" "Botswana" ...
$ obstype_year: chr "gdpPercap_1952" "gdpPercap_1952" "gdpPercap_1952" "gdpPercap_1952" ...
$ obs_values : num 2449 3521 1063 851 543 ...Eso puede parecer trivial con este data frame en
particular, pero a veces tienes una variable de identificación
ID y 40 variables de observación, con varios nombres de
variables irregulares. La flexibilidad que nos da tidyr ¡es
un gran ahorro de tiempo!
Ahora, obstype_year en realidad contiene información en
dos partes, la observación tipo (pop,lifeExp,
o gdpPercap) y el año year. Podemos usar la
función separate() para dividir las cadenas de caracteres
en múltiples variables.
R
gap_long <- gap_long %>% separate(obstype_year, into = c("obs_type", "year"), sep = "_")
gap_long$year <- as.integer(gap_long$year)
Desafío 2
Usando el data frame gap_long, calcula
el promedio de esperanza de vida, población, y PIB por cada continente.
Ayuda: usa group_by() y
summarize() que son las funciones de dplyr que
aprendiste en el episodio anterior.
R
gap_long %>%
group_by(continent, obs_type) %>%
summarize(means = mean(obs_values))
SALIDA
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.SALIDA
# A tibble: 15 × 3
# Groups: continent [5]
continent obs_type means
<chr> <chr> <dbl>
1 Africa gdpPercap 2194.
2 Africa lifeExp 48.9
3 Africa pop 9916003.
4 Americas gdpPercap 7136.
5 Americas lifeExp 64.7
6 Americas pop 24504795.
7 Asia gdpPercap 7902.
8 Asia lifeExp 60.1
9 Asia pop 77038722.
10 Europe gdpPercap 14469.
11 Europe lifeExp 71.9
12 Europe pop 17169765.
13 Oceania gdpPercap 18622.
14 Oceania lifeExp 74.3
15 Oceania pop 8874672. Del formato largo a intermedio usando spread()
Siempre es bueno detenerse y verificar el trabajo. Entonces, usemos
el opuesto de gather() para separar nuestras variables de
observación con la función spread(). Para expandir nuestro
objeto gap_long() al formato intermedio original o al
formato ancho usaremos esta nueva función. Comencemos con el formato
intermedio.
R
gap_normal <- gap_long %>% spread(obs_type,obs_values)
dim(gap_normal)
SALIDA
[1] 1704 6R
dim(gapminder)
SALIDA
[1] 1704 6R
names(gap_normal)
SALIDA
[1] "continent" "country" "year" "gdpPercap" "lifeExp" "pop" R
names(gapminder)
SALIDA
[1] "country" "year" "pop" "continent" "lifeExp" "gdpPercap"Ahora tenemos un marco de datos intermedio gap_normal
con las mismas dimensiones que el gapminder original, pero
el orden de las variables es diferente. Arreglemos eso antes de
comprobar si son iguales con la función all.equal().
R
gap_normal <- gap_normal[,names(gapminder)]
all.equal(gap_normal,gapminder)
SALIDA
[1] "Component \"country\": 1704 string mismatches"
[2] "Component \"pop\": Mean relative difference: 1.634504"
[3] "Component \"continent\": 1212 string mismatches"
[4] "Component \"lifeExp\": Mean relative difference: 0.203822"
[5] "Component \"gdpPercap\": Mean relative difference: 1.162302"R
head(gap_normal)
SALIDA
country year pop continent lifeExp gdpPercap
1 Algeria 1952 9279525 Africa 43.077 2449.008
2 Algeria 1957 10270856 Africa 45.685 3013.976
3 Algeria 1962 11000948 Africa 48.303 2550.817
4 Algeria 1967 12760499 Africa 51.407 3246.992
5 Algeria 1972 14760787 Africa 54.518 4182.664
6 Algeria 1977 17152804 Africa 58.014 4910.417R
head(gapminder)
SALIDA
country year pop continent lifeExp gdpPercap
1 Afghanistan 1952 8425333 Asia 28.801 779.4453
2 Afghanistan 1957 9240934 Asia 30.332 820.8530
3 Afghanistan 1962 10267083 Asia 31.997 853.1007
4 Afghanistan 1967 11537966 Asia 34.020 836.1971
5 Afghanistan 1972 13079460 Asia 36.088 739.9811
6 Afghanistan 1977 14880372 Asia 38.438 786.1134Ya casi, el data frame original está ordenado por
country, continent, y year.
Entonces probemos con la función arrange().
R
gap_normal <- gap_normal %>% arrange(country, continent, year)
all.equal(gap_normal, gapminder)
SALIDA
[1] TRUE¡Muy bien! Pasamos del formato largo al intermedio y no hemos tenido ningún error en nuestro código.
Ahora pasemos a convertir del formato largo al ancho. En el formato
ancho, nosotros mantendremos el país y el continente como variables de
ID y esto va expandir las observaciones en las tres métricas
(pop,lifeExp, gdpPercap) y año
(year). Primero nosotros necesitamos crear etiquetas
apropiadas para todas nuestras nuevas variables (combinaciones de
métricas*año) y también necesitamos unificar nuestras variables de
ID para simplificar el proceso de definir el nuevo
objeto gap_wide.
R
gap_temp <- gap_long %>% unite(var_ID, continent, country, sep = "_")
str(gap_temp)
SALIDA
'data.frame': 5112 obs. of 4 variables:
$ var_ID : chr "Africa_Algeria" "Africa_Angola" "Africa_Benin" "Africa_Botswana" ...
$ obs_type : chr "gdpPercap" "gdpPercap" "gdpPercap" "gdpPercap" ...
$ year : int 1952 1952 1952 1952 1952 1952 1952 1952 1952 1952 ...
$ obs_values: num 2449 3521 1063 851 543 ...R
gap_temp <- gap_long %>%
unite(ID_var, continent, country, sep = "_") %>%
unite(var_names, obs_type, year, sep = "_")
str(gap_temp)
SALIDA
'data.frame': 5112 obs. of 3 variables:
$ ID_var : chr "Africa_Algeria" "Africa_Angola" "Africa_Benin" "Africa_Botswana" ...
$ var_names : chr "gdpPercap_1952" "gdpPercap_1952" "gdpPercap_1952" "gdpPercap_1952" ...
$ obs_values: num 2449 3521 1063 851 543 ...Usando la función unite() tenemos ahora un único
ID que es la combinación de continent,
country, y así definimos nuestras nuevas variables. Ahora
podemos usar ese resultado con la función spread().
R
gap_wide_new <- gap_long %>%
unite(ID_var, continent, country, sep = "_") %>%
unite(var_names, obs_type, year,sep = "_") %>%
spread(var_names, obs_values)
str(gap_wide_new)
SALIDA
'data.frame': 142 obs. of 37 variables:
$ ID_var : chr "Africa_Algeria" "Africa_Angola" "Africa_Benin" "Africa_Botswana" ...
$ gdpPercap_1952: num 2449 3521 1063 851 543 ...
$ gdpPercap_1957: num 3014 3828 960 918 617 ...
$ gdpPercap_1962: num 2551 4269 949 984 723 ...
$ gdpPercap_1967: num 3247 5523 1036 1215 795 ...
$ gdpPercap_1972: num 4183 5473 1086 2264 855 ...
$ gdpPercap_1977: num 4910 3009 1029 3215 743 ...
$ gdpPercap_1982: num 5745 2757 1278 4551 807 ...
$ gdpPercap_1987: num 5681 2430 1226 6206 912 ...
$ gdpPercap_1992: num 5023 2628 1191 7954 932 ...
$ gdpPercap_1997: num 4797 2277 1233 8647 946 ...
$ gdpPercap_2002: num 5288 2773 1373 11004 1038 ...
$ gdpPercap_2007: num 6223 4797 1441 12570 1217 ...
$ lifeExp_1952 : num 43.1 30 38.2 47.6 32 ...
$ lifeExp_1957 : num 45.7 32 40.4 49.6 34.9 ...
$ lifeExp_1962 : num 48.3 34 42.6 51.5 37.8 ...
$ lifeExp_1967 : num 51.4 36 44.9 53.3 40.7 ...
$ lifeExp_1972 : num 54.5 37.9 47 56 43.6 ...
$ lifeExp_1977 : num 58 39.5 49.2 59.3 46.1 ...
$ lifeExp_1982 : num 61.4 39.9 50.9 61.5 48.1 ...
$ lifeExp_1987 : num 65.8 39.9 52.3 63.6 49.6 ...
$ lifeExp_1992 : num 67.7 40.6 53.9 62.7 50.3 ...
$ lifeExp_1997 : num 69.2 41 54.8 52.6 50.3 ...
$ lifeExp_2002 : num 71 41 54.4 46.6 50.6 ...
$ lifeExp_2007 : num 72.3 42.7 56.7 50.7 52.3 ...
$ pop_1952 : num 9279525 4232095 1738315 442308 4469979 ...
$ pop_1957 : num 10270856 4561361 1925173 474639 4713416 ...
$ pop_1962 : num 11000948 4826015 2151895 512764 4919632 ...
$ pop_1967 : num 12760499 5247469 2427334 553541 5127935 ...
$ pop_1972 : num 14760787 5894858 2761407 619351 5433886 ...
$ pop_1977 : num 17152804 6162675 3168267 781472 5889574 ...
$ pop_1982 : num 20033753 7016384 3641603 970347 6634596 ...
$ pop_1987 : num 23254956 7874230 4243788 1151184 7586551 ...
$ pop_1992 : num 26298373 8735988 4981671 1342614 8878303 ...
$ pop_1997 : num 29072015 9875024 6066080 1536536 10352843 ...
$ pop_2002 : num 31287142 10866106 7026113 1630347 12251209 ...
$ pop_2007 : num 33333216 12420476 8078314 1639131 14326203 ...Desafío 3
Crea un formato de datos gap_super_wide mediante la
distribución por países, año y las tres métricas. Ayuda
este nuevo data frame sólo debe tener cinco filas.
R
gap_super_wide <- gap_long %>%
unite(var_names, obs_type, year, country, sep = "_") %>%
spread(var_names, obs_values)
Ahora tenemos un gran data frame con formato
‘ancho’, pero el ID_var podría ser más mejor, hay que
separarlos en dos variables con separate()
R
gap_wide_betterID <- separate(gap_wide_new, ID_var, c("continent", "country"), sep = "_")
gap_wide_betterID <- gap_long %>%
unite(ID_var, continent, country, sep = "_") %>%
unite(var_names, obs_type, year, sep = "_") %>%
spread(var_names, obs_values) %>%
separate(ID_var, c("continent", "country"), sep = "_")
str(gap_wide_betterID)
SALIDA
'data.frame': 142 obs. of 38 variables:
$ continent : chr "Africa" "Africa" "Africa" "Africa" ...
$ country : chr "Algeria" "Angola" "Benin" "Botswana" ...
$ gdpPercap_1952: num 2449 3521 1063 851 543 ...
$ gdpPercap_1957: num 3014 3828 960 918 617 ...
$ gdpPercap_1962: num 2551 4269 949 984 723 ...
$ gdpPercap_1967: num 3247 5523 1036 1215 795 ...
$ gdpPercap_1972: num 4183 5473 1086 2264 855 ...
$ gdpPercap_1977: num 4910 3009 1029 3215 743 ...
$ gdpPercap_1982: num 5745 2757 1278 4551 807 ...
$ gdpPercap_1987: num 5681 2430 1226 6206 912 ...
$ gdpPercap_1992: num 5023 2628 1191 7954 932 ...
$ gdpPercap_1997: num 4797 2277 1233 8647 946 ...
$ gdpPercap_2002: num 5288 2773 1373 11004 1038 ...
$ gdpPercap_2007: num 6223 4797 1441 12570 1217 ...
$ lifeExp_1952 : num 43.1 30 38.2 47.6 32 ...
$ lifeExp_1957 : num 45.7 32 40.4 49.6 34.9 ...
$ lifeExp_1962 : num 48.3 34 42.6 51.5 37.8 ...
$ lifeExp_1967 : num 51.4 36 44.9 53.3 40.7 ...
$ lifeExp_1972 : num 54.5 37.9 47 56 43.6 ...
$ lifeExp_1977 : num 58 39.5 49.2 59.3 46.1 ...
$ lifeExp_1982 : num 61.4 39.9 50.9 61.5 48.1 ...
$ lifeExp_1987 : num 65.8 39.9 52.3 63.6 49.6 ...
$ lifeExp_1992 : num 67.7 40.6 53.9 62.7 50.3 ...
$ lifeExp_1997 : num 69.2 41 54.8 52.6 50.3 ...
$ lifeExp_2002 : num 71 41 54.4 46.6 50.6 ...
$ lifeExp_2007 : num 72.3 42.7 56.7 50.7 52.3 ...
$ pop_1952 : num 9279525 4232095 1738315 442308 4469979 ...
$ pop_1957 : num 10270856 4561361 1925173 474639 4713416 ...
$ pop_1962 : num 11000948 4826015 2151895 512764 4919632 ...
$ pop_1967 : num 12760499 5247469 2427334 553541 5127935 ...
$ pop_1972 : num 14760787 5894858 2761407 619351 5433886 ...
$ pop_1977 : num 17152804 6162675 3168267 781472 5889574 ...
$ pop_1982 : num 20033753 7016384 3641603 970347 6634596 ...
$ pop_1987 : num 23254956 7874230 4243788 1151184 7586551 ...
$ pop_1992 : num 26298373 8735988 4981671 1342614 8878303 ...
$ pop_1997 : num 29072015 9875024 6066080 1536536 10352843 ...
$ pop_2002 : num 31287142 10866106 7026113 1630347 12251209 ...
$ pop_2007 : num 33333216 12420476 8078314 1639131 14326203 ...¡Muy bien hiciste el cambio de formato de ida y vuelta!
Otros recursos geniales
- [R para Ciencia de datos] (r4ds.had.co.nz)
- [Hoja de trucos para la conversión de datos] (https://www.rstudio.com/wp-content/uploads/2015/02/data-wrangling-cheatsheet.pdf)
- [Introducción a tidyr] (https://cran.r-project.org/web/packages/tidyr/vignettes/tidy-data.html)
- [Manipulación de datos con R y RStudio] (https://www.rstudio.com/resources/webinars/data-wrangling-with-r-and-rstudio/)
- Usar el paquete
tidyrpara cambiar el diseño de los data frames. - Usar
gather()para invertir del formato ancho al formato largo. - Usar
spread()para invertir del formato largo al formato ancho.
