Manipulación de data frames con dplyr
Última actualización: 2025-11-11 | Mejora esta página
Hoja de ruta
Preguntas
- ¿Cómo manipular data frames sin repetir lo mismo una y otra vez?
Objetivos
- Ser capaces de usar las seis principales acciones de manipulación de
data frames con pipes en
dplyr. - Comprender cómo combinar
group_by()ysummarize()para obtener resúmenes de datasets. - Ser capaces de analizar un subconjunto de datos usando un filtrado lógico.
Palabras clave
Comando : Traducción
filter : filtrar
select : seleccionar
group_by : agrupar
summarize : resumir
count : contar
mean : media
mutate : mutar
La manipulación de data frames significa distintas cosas para distintos investigadores. A veces queremos seleccionar ciertas observaciones (filas) o variables (columnas), otras veces deseamos agrupar los datos en función de una o más variables, o queremos calcular valores estadísticos de un conjunto. Podemos hacer todo ello usando las habituales operaciones básicas de R:
R
mean(gapminder[gapminder$continent == "Africa", "gdpPercap"])
SALIDA
[1] 2193.755R
mean(gapminder[gapminder$continent == "Americas", "gdpPercap"])
SALIDA
[1] 7136.11R
mean(gapminder[gapminder$continent == "Asia", "gdpPercap"])
SALIDA
[1] 7902.15Pero esto no es muy elegante porque hay demasiada repetición. Repetir cosas cuesta tiempo, tanto en el momento de hacerlo como en el futuro, y aumenta la probabilidad de que se produzcan desagradables bugs (errores).
El paquete dplyr
Afortunadamente, el paquete dplyr
proporciona un conjunto de funciones (consulta la guía
rápida ) extremadamente útiles para manipular data
frames y así reducir el número de repeticiones , la
probabilidad de cometer errores y el número de caracteres que hay que
escribir. Como valor extra, puedes encontrar que la gramática de
dplyr es más fácil de entender.
Aquí vamos a revisar 6 de sus funciones más usadas, así como a usar
los pipes (%>%) para combinarlas.
select()filter()group_by()summarize()mutate()
Si no has instalado antes este paquete, hazlo del siguiente modo:
R
install.packages('dplyr')
Ahora vamos a cargar el paquete:
R
library("dplyr")
Usando select()
Si por ejemplo queremos continuar el trabajo con sólo unas pocas de
las variables de nuestro data frame podemos usar la
función select(). Esto guardará sólo las variables que
seleccionemos.
R
year_country_gdp <- select(gapminder,year,country,gdpPercap)
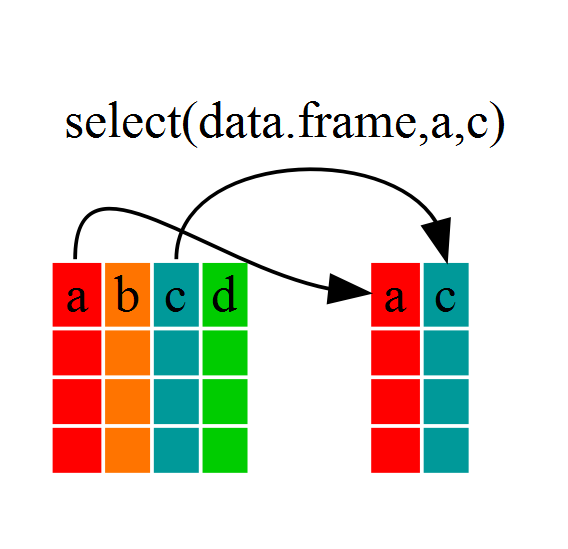
Si ahora investigamos year_country_gdp veremos que sólo
contiene el año, el país y la renta per cápita. Arriba hemos usado la
gramática ‘normal’, pero la fortaleza de dplyr consiste en
combinar funciones usando pipes. Como la gramática de
las pipes es distinta a todo lo que hemos visto antes
en R, repitamos lo que hemos hecho arriba, pero esta vez usando
pipes.
R
year_country_gdp <- gapminder %>% select(year,country,gdpPercap)
Para ayudarte a entender por qué lo hemos escrito así, vamos a
revisarlo por partes. Primero hemos llamado al data
frame “gapminder” y se lo hemos pasado al siguiente paso, que
es la función select(), usando el símbolo del
pipe %>%. En este caso no especificamos
qué objeto de datos vamos a usar en la función select()
porque esto se obtiene del resultado de la instrucción previa a el
pipe. Dato curioso: es muy posible que
te hayas encontrado con pipes antes en la terminal de
unix. En R el símbolo del pipe es %>%,
mientras que en la terminal es |, pero el concepto es el
mismo.
Usando filter()
Si ahora queremos continuar con lo de arriba, pero sólo con los
países europeos, podemos combinar select y
filter.
R
year_country_gdp_euro <- gapminder %>%
filter(continent=="Europe") %>%
select(year,country,gdpPercap)
Reto 1
Escribe un único comando (que puede ocupar varias líneas e incluir
pipes) que produzca un data frame y
que tenga los valores africanos correspondientes a lifeExp,
country y year, pero no de los otros
continentes. ¿Cuántas filas tiene dicho data frame y
por qué?
R
year_country_lifeExp_Africa <- gapminder %>%
filter(continent=="Africa") %>%
select(year,country,lifeExp)
Al igual que la vez anterior, primero le pasamos el data
frame “gapminder” a la función filter() y luego le
pasamos la versión filtrada del data frame a la función
select(). Nota: El orden de las
operaciones es muy importante en este caso. Si usamos primero
select(), la función filter() no habría podido
encontrar la variable “continent” porque la habríamos eliminado en el
paso previo.
Usando group_by() y summarize()
Se suponía que teníamos que reducir las repeticiones causantes de
errores de lo que se puede hacer con el R básico, pero hasta ahora no lo
hemos conseguido porque tendríamos que repetir lo escrito arriba para
cada continente. En lugar de filter(), que solamente deja
pasar las observaciones que se ajustan a tu criterio
(continent = Europe en lo escrito arriba), podemos usar
group_by(), que esencialmente usará cada uno de los
criterios únicos que podrías haber usado con filter().
R
str(gapminder)
SALIDA
'data.frame': 1704 obs. of 6 variables:
$ country : chr "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num 779 821 853 836 740 ...R
str(gapminder %>% group_by(continent))
SALIDA
gropd_df [1,704 × 6] (S3: grouped_df/tbl_df/tbl/data.frame)
$ country : chr [1:1704] "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int [1:1704] 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num [1:1704] 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr [1:1704] "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num [1:1704] 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num [1:1704] 779 821 853 836 740 ...
- attr(*, "groups")= tibble [5 × 2] (S3: tbl_df/tbl/data.frame)
..$ continent: chr [1:5] "Africa" "Americas" "Asia" "Europe" ...
..$ .rows : list<int> [1:5]
.. ..$ : int [1:624] 25 26 27 28 29 30 31 32 33 34 ...
.. ..$ : int [1:300] 49 50 51 52 53 54 55 56 57 58 ...
.. ..$ : int [1:396] 1 2 3 4 5 6 7 8 9 10 ...
.. ..$ : int [1:360] 13 14 15 16 17 18 19 20 21 22 ...
.. ..$ : int [1:24] 61 62 63 64 65 66 67 68 69 70 ...
.. ..@ ptype: int(0)
..- attr(*, ".drop")= logi TRUESe puede observar que la estructura del data frame
obtenido por group_by() (grouped_df) no es la
misma que la del data frame original
gapminder(data.fram). Se puede pensar en un
grouped_df como en una list donde cada item in
la list es un data.frame que contiene
únicamente las filas que corresponden a un valor particular de
continent (en el ejemplo mostrado).
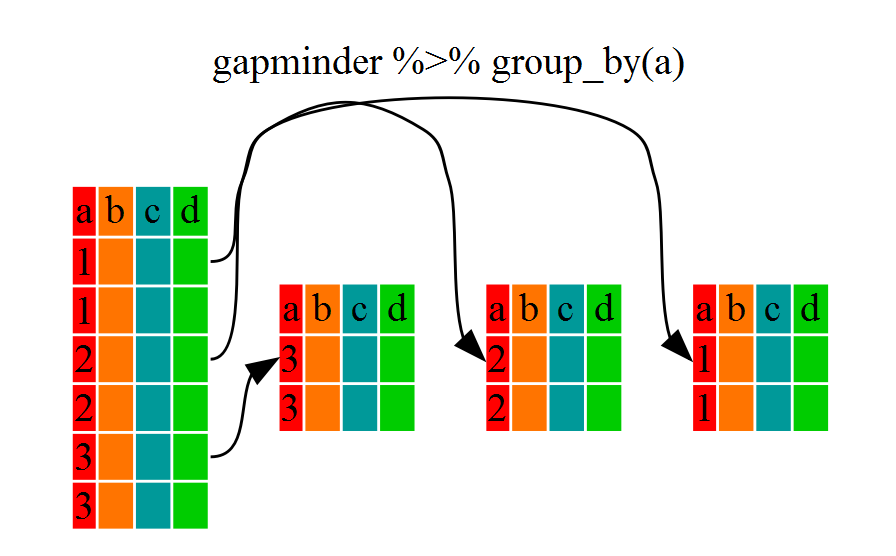
Usando summarize()
Lo visto arriba no es muy sofisticado, pero group_by()
es más interesante y útil si se usa en conjunto con
summarize(). Esto nos permitirá crear nuevas variables
usando funciones que se aplican a cada uno de los data
frames específicos para cada continente. Es decir, usando la
función group_by() dividimos nuestro data
frame original en varias partes, a las que luego podemos
aplicarles funciones (por ejemplo, mean() o
sd()) independientemente con summarize().
R
gdp_bycontinents <- gapminder %>%
group_by(continent) %>%
summarize(mean_gdpPercap=mean(gdpPercap))
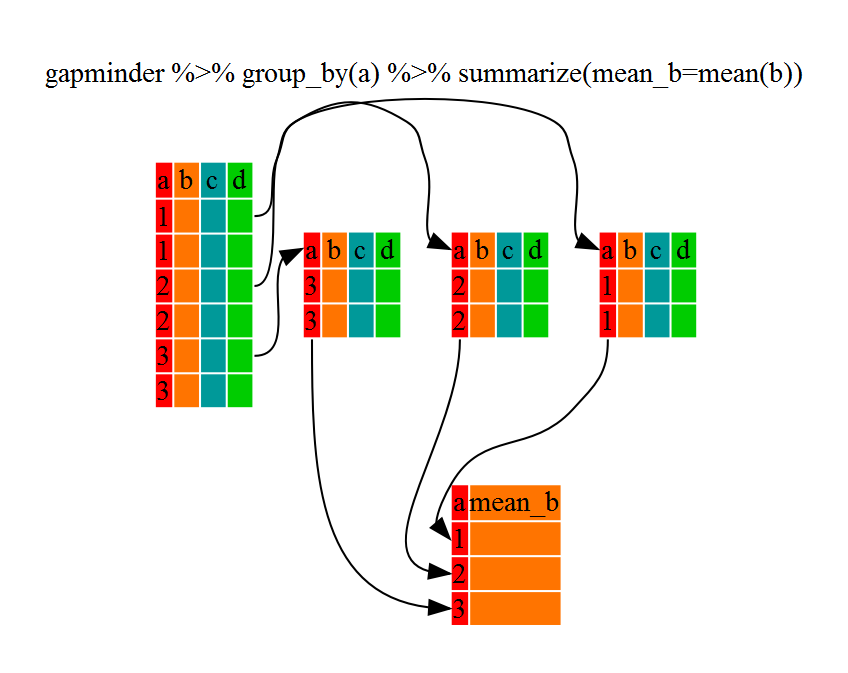
R
continent mean_gdpPercap
<fctr> <dbl>
1 Africa 2193.755
2 Americas 7136.110
3 Asia 7902.150
4 Europe 14469.476
5 Oceania 18621.609Esto nos ha permitido calcular la renta per cápita media para cada continente, pero puede ser todavía mucho mejor.
Reto 2
Calcula la esperanza de vida media por país. ¿Qué país tiene la esperanza de vida media mayor y cuál la menor?
R
lifeExp_bycountry <- gapminder %>%
group_by(country) %>%
summarize(mean_lifeExp=mean(lifeExp))
lifeExp_bycountry %>%
filter(mean_lifeExp == min(mean_lifeExp) | mean_lifeExp == max(mean_lifeExp))
SALIDA
# A tibble: 2 × 2
country mean_lifeExp
<chr> <dbl>
1 Iceland 76.5
2 Sierra Leone 36.8Otro modo de hacer esto es usando la función arrange()
del paquete dplyr, que distribuye las filas de un
data frame en función del orden de una o más variables
del data frame. Tiene una sintaxis similar a otras
funciones del paquete dplyr. Se puede usar
desc() dentro de arrange() para ordenar de
modo descendente.
R
lifeExp_bycountry %>%
arrange(mean_lifeExp) %>%
head(1)
SALIDA
# A tibble: 1 × 2
country mean_lifeExp
<chr> <dbl>
1 Sierra Leone 36.8R
lifeExp_bycountry %>%
arrange(desc(mean_lifeExp)) %>%
head(1)
SALIDA
# A tibble: 1 × 2
country mean_lifeExp
<chr> <dbl>
1 Iceland 76.5La función group_by() nos permite agrupar en función de
varias variables. Vamos a agrupar por year y
continent.
R
gdp_bycontinents_byyear <- gapminder %>%
group_by(continent,year) %>%
summarize(mean_gdpPercap=mean(gdpPercap))
SALIDA
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.Esto ya es bastante potente, pero puede ser incluso mejor. Puedes
definir más de una variable en summarize().
R
gdp_pop_bycontinents_byyear <- gapminder %>%
group_by(continent,year) %>%
summarize(mean_gdpPercap=mean(gdpPercap),
sd_gdpPercap=sd(gdpPercap),
mean_pop=mean(pop),
sd_pop=sd(pop))
SALIDA
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.count() y n()
Una operación muy habitual es contar el número de observaciones que
hay en cada grupo. El paquete dplyr tiene dos funciones
relacionadas muy útiles para ello.
Por ejemplo, si queremos comprobar el número de países que hay en el
conjunto de datos para el año 2002 podemos usar la función
count(). Dicha función toma el nombre de una o más columnas
que contienen los grupos en los que estamos interesados y puede
opcionalmente ordenar los resultados en modo descendente si añadimos
sort = TRUE.
R
gapminder %>%
filter(year == 2002) %>%
count(continent, sort = TRUE)
SALIDA
continent n
1 Africa 52
2 Asia 33
3 Europe 30
4 Americas 25
5 Oceania 2Si necesitamos usar en nuestros cálculos el número de observaciones
obtenidas, la función n() es muy útil. Por ejemplo, si
queremos obtener el error estándar de la esperanza de vida por
continente:
R
gapminder %>%
group_by(continent) %>%
summarize(se_pop = sd(lifeExp)/sqrt(n()))
SALIDA
# A tibble: 5 × 2
continent se_pop
<chr> <dbl>
1 Africa 0.366
2 Americas 0.540
3 Asia 0.596
4 Europe 0.286
5 Oceania 0.775También se pueden encadenar juntas varias operaciones de resumen,
como en el caso siguiente, en el que calculamos el minimum,
maximum, mean y se de la
esperanza de vida por país para cada continente:
R
gapminder %>%
group_by(continent) %>%
summarize(
mean_le = mean(lifeExp),
min_le = min(lifeExp),
max_le = max(lifeExp),
se_le = sd(lifeExp)/sqrt(n()))
SALIDA
# A tibble: 5 × 5
continent mean_le min_le max_le se_le
<chr> <dbl> <dbl> <dbl> <dbl>
1 Africa 48.9 23.6 76.4 0.366
2 Americas 64.7 37.6 80.7 0.540
3 Asia 60.1 28.8 82.6 0.596
4 Europe 71.9 43.6 81.8 0.286
5 Oceania 74.3 69.1 81.2 0.775Usando mutate()
También se pueden crear nuevas variables antes (o incluso después) de
resumir la información usando mutate().
R
gdp_pop_bycontinents_byyear <- gapminder %>%
mutate(gdp_billion=gdpPercap*pop/10^9) %>%
group_by(continent,year) %>%
summarize(mean_gdpPercap=mean(gdpPercap),
sd_gdpPercap=sd(gdpPercap),
mean_pop=mean(pop),
sd_pop=sd(pop),
mean_gdp_billion=mean(gdp_billion),
sd_gdp_billion=sd(gdp_billion))
SALIDA
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.Conectando mutate con filtrado lógico: ifelse
La creación de nuevas variables se puede conectar con una condición
lógica. Una simple combinación de mutate y
ifelse facilita el filtrado solo allí donde se necesita: en
el momento de crear algo nuevo. Esta combinación es fácil de leer y es
un modo rápido y potente de descartar ciertos datos (incluso sin cambiar
la dimensión conjunta del data frame) o para actualizar
valores dependiendo de la condición utilizada.
R
## manteniendo todos los datos pero "filtrando" según una determinada condición
# calcular renta per cápita sólo para gente con una esperanza de vida por encima de 25
gdp_pop_bycontinents_byyear_above25 <- gapminder %>%
mutate(gdp_billion = ifelse(lifeExp > 25, gdpPercap * pop / 10^9, NA)) %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
sd_gdpPercap = sd(gdpPercap),
mean_pop = mean(pop),
sd_pop = sd(pop),
mean_gdp_billion = mean(gdp_billion),
sd_gdp_billion = sd(gdp_billion))
SALIDA
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.R
## actualizando sólo si se cumple una determinada condición
# para esperanzas de vida por encima de 40 años, el GDP que se espera en el futuro es escalado
gdp_future_bycontinents_byyear_high_lifeExp <- gapminder %>%
mutate(gdp_futureExpectation = ifelse(lifeExp > 40, gdpPercap * 1.5, gdpPercap)) %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
mean_gdpPercap_expected = mean(gdp_futureExpectation))
SALIDA
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.Combinando dplyr y ggplot2
En la función de creación de gráficas vimos cómo hacer una figura con múltiples paneles añadiendo una capa de paneles separados (facet panels). Aquí está el código que usamos (con algunos comentarios extra):
R
# Obtener la primera letra de cada país
starts.with <- substr(gapminder$country, start = 1, stop = 1)
# Filtrar países que empiezan con "A" o "Z"
az.countries <- gapminder[starts.with %in% c("A", "Z"), ]
# Construir el gráfico
ggplot(data = az.countries, aes(x = year, y = lifeExp, color = continent)) +
geom_line() + facet_wrap( ~ country)
ERROR
Error in ggplot(data = az.countries, aes(x = year, y = lifeExp, color = continent)): could not find function "ggplot"Este código construye la gráfica correcta, pero también crea algunas
variables starts.with y az.countries) que
podemos no querer usar para nada más. Del mismo modo que usamos
%>% para pasar datos con pipes a lo
largo de una cadena de funciones dplyr, podemos usarlo para
pasarle datos a ggplot(). Como %>%
sustituye al primer argumento de una función, no necesitamos especificar
el argumento data= de la función ggplot().
Combinando funciones de los paquetes dplyr y
ggplot podemos hacer la misma figura sin crear ninguna
nueva variable y sin modificar los datos.
R
gapminder %>%
# Get the start letter of each country
mutate(startsWith = substr(country, start = 1, stop = 1)) %>%
# Filter countries that start with "A" or "Z"
filter(startsWith %in% c("A", "Z")) %>%
# Make the plot
ggplot(aes(x = year, y = lifeExp, color = continent)) +
geom_line() +
facet_wrap( ~ country)
ERROR
Error in ggplot(., aes(x = year, y = lifeExp, color = continent)): could not find function "ggplot"Las funciones del paquete dplyr también nos ayudan a
simplificar las cosas, por ejemplo, combinando los primeros dos
pasos:
R
gapminder %>%
# Filter countries that start with "A" or "Z"
filter(substr(country, start = 1, stop = 1) %in% c("A", "Z")) %>%
# Make the plot
ggplot(aes(x = year, y = lifeExp, color = continent)) +
geom_line() +
facet_wrap( ~ country)
ERROR
Error in ggplot(., aes(x = year, y = lifeExp, color = continent)): could not find function "ggplot"Reto Avanzado
Calcula la esperanza de vida media en 2002 de dos países
seleccionados al azar para cada continente. Luego distribuye los nombres
de los continentes en orden inverso. Pista: Usa las
funciones arrange() y sample_n() del paquete
dplyr, tienen una sintaxis similar a las demás funciones
del paquete dplyr.
R
lifeExp_2countries_bycontinents <- gapminder %>%
filter(year==2002) %>%
group_by(continent) %>%
sample_n(2) %>%
summarize(mean_lifeExp=mean(lifeExp)) %>%
arrange(desc(mean_lifeExp))
Más información
- R for Data Science
- Data Wrangling Cheat sheet
- Introduction to dplyr
- Data wrangling with R and RStudio
- Usar el paquete
dplyrpara manipular data frames. - Usar
select()para seleccionar variables de un data frame. - Usar
filter()para seleccionar datos basándose en los valores. - Usar
group_by()ysummarize()para trabajar con subconjuntos de datos. - Usar
mutate()para crear nuevas variables.
